遺伝子の復帰・修復
復帰遺伝子
遺伝子が変異することもあれば、変異した遺伝子が元通りに治ることもある。これが遺伝子の復帰である。
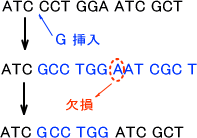
・復帰の例(Leu → Phe → Leu)
Leuをコードする配列に「TTA」がある。これがA→Tへと変異すると「TTT」となり、Pheをコードする配列に変化する。しかし、もう一度T→Aに変化すると「TTA」となりLeuのコードに戻る。

ただし、復帰変異には「塩基配列が元に戻る場合」と「元の塩基配列ではないが、コードしているアミノ酸配列が戻る場合」がある。ここでは両方とも復帰変異という。
・フレームシフトの変異
フレームシフト変異したとしても、もう一度塩基がフレームシフトして復帰することがある。ただし、この場合の復帰変異は最初に変異した部分とは違う塩基が変異する場合が多い。全く同じ塩基が復帰することはあるがその確立は低い。
フレームシフト変異の復帰変異には下のような条件がある。
・最初に変異した部分と復帰変異の場所が近い
・変異した部分がタンパク質の性質に大きな影響を与えない
・サプレッサー変異
この変異はtRNAの変異によって起こる。サプレッサー変異を起こしたtRNAはアンチコドンに対応するはずのアミノ酸が異なっているのである。
例えば、mRNAが「UUA」とコードしていたとする。これに対応するtRNAが運ぶアミノ酸はLeuである。しかし、DNAに変異が起こった結果としてmRNAのコードが「UUA」から「UUU」に変化していたとする。すると、tRNAはLeuではなくてPheを運んできてしまい変異が起こる。
もしここでサプレッサー変異が起こると、前で述べた変異が打ち消される。つまり、tRNAのアンチコドンの部分が「AAU」から「AAA」に変異するのである。アンチコドンが変異してもtRNAがもつアミノ酸は変わらない。
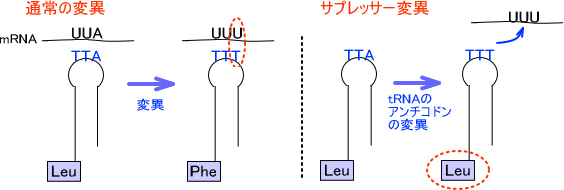
上の図の通りにmRNAが変異すると通常ならLeuではなくPheが来るはずである。しかし、Pheを入れるべきところにLeuを入れるように変異したtRNAが来ると最終的にはにはLeuが入るので、見かけ上は復帰していることになる。
損傷の修復
・直接修復
DNA上にチミンが二つ並んでいるとき、紫外線を受けるとチミン二量体を形成してしまう。このチミン二量体は光回復酵素によって修復される。光回復酵素は可視光によって活性化する。

・アルキル化の修復
O6-メチルグアニンやO4-メチルチミンなどのアルキル化の修復はO6-メチルグアニン-DNA-メチルトランスフェラーゼ(MGT)によって行われる。
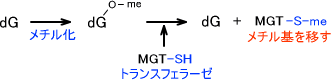
MGTはアルキル化した塩基にあるメチル基を自分に移す。こうすることによって塩基のアルキル化を修復する。ただし、これによって酵素は失活するのでMGTは自殺酵素と呼ばれている。
損傷除去修復
・塩基除去修復
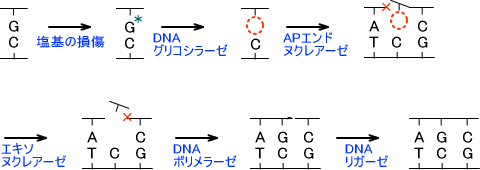
これは塩基が損傷した部分を酵素によって切り取って再びつなぎ合わせる方法である。1塩基を切り取る場合はDNAグリコシラーゼによって行われる。DNAグリコシラーゼは損傷塩基を外し、APサイトを作る働きをする。
APサイトが作られると、APエンドヌクレアーゼによってAPサイトが存在する部分が1ヶ所切断される。その後、エキソヌクレアーゼによってAPサイトは完全に除去される。
APサイトが除去されるとDNAポリメラーゼによって新しく塩基が作られる。そして、最後にDNAリガーゼがニックを埋めて修復が完了する。
DNA中のグアニンが酸化されて8-オキソグアニンができた場合、ヒトでこの8-オキソグアニンを取り除くDNAグリコシラーゼをOGG1という。大腸菌でこの働きをする酵素にFapyやmutMがある。
また、DNA中の塩基ではアデニンも酸化される。アデニンが酸化されると2-ヒドロキシアデニンとなる。ヒトで2-ヒドロキシアデニンを取り除く働きをする酵素はMUTYH1であり、大腸菌ではmutYである。
・ヌクレオチド除去修復
チミン二量体を修復するとき、光回復を利用しない修復の仕方が存在する。この方法は酵素によって行われる。
塩基を切り取るとき、チミン二量体を挟んで12~13ヌクレオチドで切り取る。大腸菌ではUVエンドヌクレアーゼによって切り取られる。その後、DNAポリメラーゼとDNAリガーゼの働きによって修復が完了する。
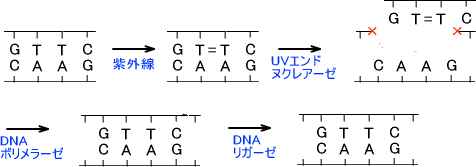
遺伝性の病気に除去修復遺伝子欠損症がある。この病気は色素性乾皮症といい、チミン二量体をDNAから除去する能力が減少している。
色素性乾皮症の人は紫外線による障害を受けやすく、少量の日光を浴びただけで皮膚がんが多発してしまう。
酸化されたdGTの排除
dGTPはDNA中のグアニン塩基の原料として、DNA複製の時にポリメラーゼが利用する。塩基が酸化されるのはDNA中の塩基だけでなく、このdGTPが酸化されることもある。
dGTPが酸化されると8-オキソグアニンとなる。つまり、8-オキソグアニンとしてポリメラーゼに利用されるのである。8-オキソグアニンはシトシン以外にもアデニンとも対合する。
これを阻止するために、酸化されたdGTPを使えない形にする酵素がある。この酵素はdGTPをdGMPに変化させる。dGMPにまで変化されると再びdGTPに戻ることはなく、そのまま排泄される。ヒトでこの働きをする酵素をMTH1といい、大腸菌の場合はmutTという。
dATP → (酸化) → 8-oxo-dGTP → (MUTYH1、mutY) → 8-oxo-dGMP → 排泄
複製後修復
・ミスマッチ修復
DNAポリメラーゼは塩基を挿入するときに間違いを起こすことがある。間違いはすぐに訂正されるが、間違いが見逃されることがある。このように訂正されなかった塩基にはミスマッチ修復(不適合修復)という機構が働いて修復される。
塩基のエラーが確認されたとき、どちらが親鎖かを見分ける必要がある。見分けるときにはアデニンのメチル化で親鎖を決定する。親鎖のところどことには-GATC-配列があり、この配列中のアデニンはメチル化されているのである。

エラーを修正するためにヌクレオチドを切断するとき、まずどちらが親鎖かを認識する。その後、エラー箇所から近い方のメチル化部位の反対側を切る。ヌクレオチドが取り除かれる部分はエラー部分からメチル化部分までである。
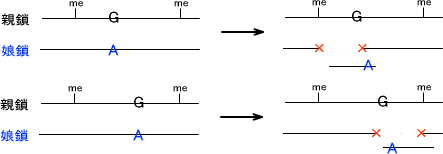
切断した後はDNAポリメラーゼ、DNAリガーゼによって再合成される。
また、「どのようにしてミスマッチ部位から近いメチル部分を見分けるか」であるが、まずループを作ってミスマッチ修復タンパクがミスマッチ部位からだんだんと下がっていき、メチル化部位を見つけたらそこで切断すると考えられている。

・組み換え修復
変異が修復されないまま複製が始まると、とても都合が悪いことが起こる。例えば、チミン二量体にポリメラーゼが当たると、複製がストップしてしまう。しかし、複製を止めるわけにはいかないので修復機構が働いて、DNAを修復しようとする。
組み換え修復では、損傷のあるDNAの反対側の鎖を利用する。つまり、損傷していない方のDNAを切り取って貼り付けし、再び合成するのである。
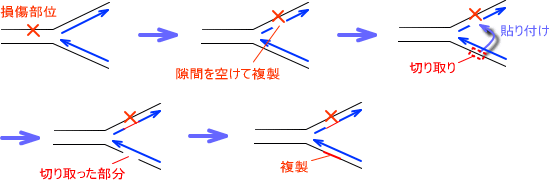
複製するときに損傷部位があると、その部位を空けて複製する。その後、損傷のない反対側のDNAを切り取って、空けて複製した部位に貼り付ける。すると、切り取った部位に隙間ができるので、今度は複製したばかりのDNA(上の図では青色のDNA)を鋳型にして、切り取った部分を複製する。
・SOS応答
SOS修復は大腸菌で有名である。DNA上に多数の傷ができた場合、それ以上のDNA合成が進まなくなってしまう。しかし、このままではその大腸菌は死んでしまう。これを回避するのがSOS応答である。
SOS応答ではポリメラーゼⅢの校正機能を抑制する。これによって、無理やりDNA合成を進めるのである。この方法は当然であるが、変異が起きやすい。しかし、大腸菌は死なずにすむ。
スポンサードリンク
カテゴリー
スポンサードリンク
